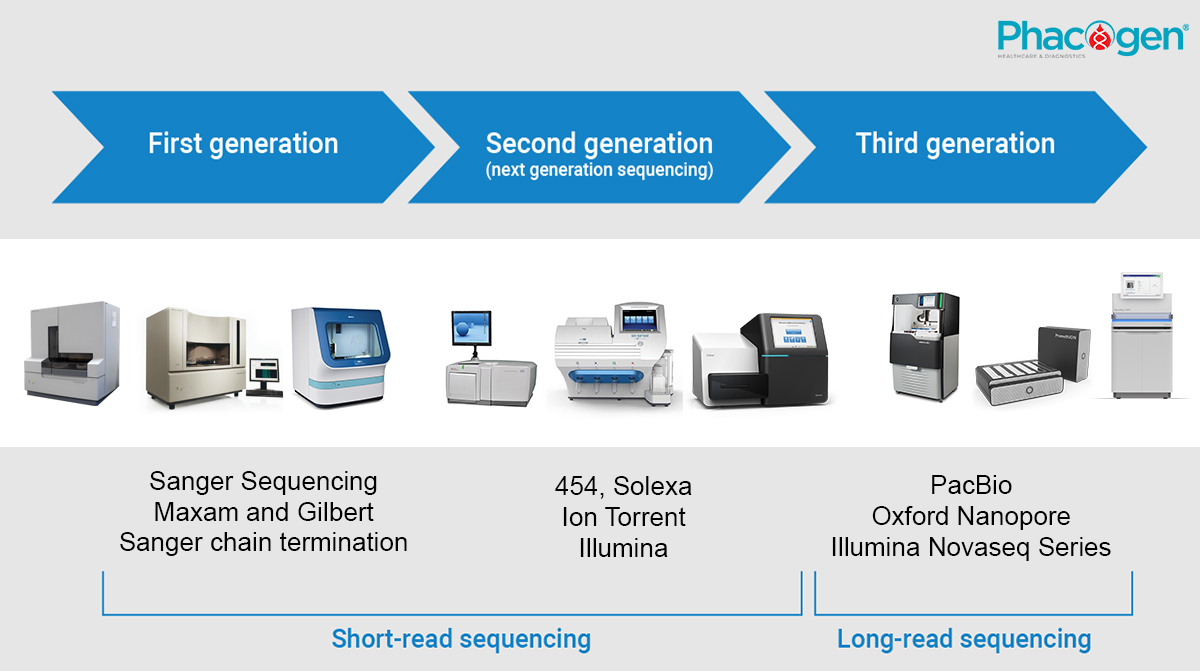
Giải trình tự thế hệ thứ nhất — bắt đầu kỷ nguyên của bộ gen
Phương pháp giải trình tự Sanger là phương pháp giải mã chuỗi DNA thế hệ đầu tiên, được phát triển bởi Frederick Sanger và các cộng sự vào năm 1977. Vào những năm 1980, phương pháp gốc của Sanger được tự động hóa bởi các nhà khoa học tại Caltech và thương mại hóa bởi Applied Biosystems.
4 ống nghiệm chứa 4 loại ddNTP khác nhau được đánh dấu đồng vị phóng xạ đồng thời được thực hiện phản ứng tổng hợp. Sau đó sản phẩm của 4 ống nghiệm được điện di hiện hình đồng vị phóng xạ trên cùng một bản gel để quan sát các band DNA. Căn cứ vào vị trí các vạch quan sát được trên bản gel mà xác định trình tự các nucleotide theo chiều từ 5 -3 từ dưới lên trên.
Ngày nay người ta sử dụng các máy giải trình tự động để sắp xếp trình tự các band điện di. Kỹ thuật giải trình tự DNA tự động (Dye temination sequencing) sử dụng các ddNTP có đánh dấu huỳnh quang giúp cho việc sắp xếp trình tự DNA trở nên nhanh chóng và hiệu quả hơn. Mỗi loại ddNTP được gắn một màu có bước sóng phát xạ huỳnh quang khác nhau cho phép thực hiện giải trình tự trong một phản ứng. Hiện nay, công nghệ giải trình Sanger tự động vẫn được sử dụng, chủ yếu trong các phòng thí nghiệm lâm sàng-nơi có thể chấp nhận tỷ lệ sản phẩm thấp, chi phí cao hơn cho mỗi mẫu và đọc chuỗi dài từ 500-1.000 bp.
Giải trình tự thế hệ thứ hai – short read (đọc dữ liệu ngắn) trở nên nhanh chóng và hiệu quả
Illumina sau khi mua lại công ty Solexa đã phát triển mạnh mẽ công nghệ giải trình tự gen thế hệ mới (NGS). Điểm mấu chốt của công nghệ NGS trên nền tảng của Illumina là " bridge amplification " (tăng cầu nối độ khuếch đại), cho phép tạo thành các cụm dày đặc các đoạn tăng tưởng trên một chip silicon. Giải trình tự dựa trên nguyên lý tổng hợp (SBS- Sequencing by synthesis), từ phân tử đơn ban đầu được khuếch đại thành một cụm lớn nhiều bản sao, đủ để phát hiện tín hiệu huỳnh quang khi thêm một dNTP đơn lẻ mỗi lần. Theo thời gian, số lượng cụm có thể đọc tăng lên đồng thời đáng kể, thiết bị của Illumina trở thành sản phẩm công nghệ giải trình tự song song số lượng lớn được thương mại hóa thành công. NGS là công nghệ giải trình tự được sử dụng chủ yếu hiện nay. Sức chứa cực đại của chúng cho phép giải trình tự với chi phí rất thấp. Tuy nhiên, NGS có giới hạn về độ dài đọc; các nền tảng NGS thường tạo ra các lượt đọc có độ dài khoảng 50-500 bp, phù hợp với các dự án resequencing, SNP calling và giải trình tự mục tiêu của các amplicon rất ngắn.
Trình tự thế hệ thứ ba - sự gia tăng của long read (đọc dữ liệu dài)
Đọc dữ liệu ngắn (short read) không phù hợp với tất cả các dự án giải trình tự gen. Kỹ thuật giải trình tự đơn phân tử, thời gian thực (SMRT- Single-molecule real-time sequencing) của PacBio được thực hiện trong một lỗ kích thước nano với tính chất đặc biệt: cho phép quan sát tín hiệu huỳnh quang đơn phân tử khi chất huỳnh quang tiến sát vào đáy của lỗ nano (zero-mode waveguide).
Một phương pháp đọc dữ liệu dài khác dựa trên tín hiệu điện phân tử được phát triển bởi Oxford Nanopore Technologies (ONT). Các phiên bản mới nhất của các hệ thống đọc dữ liệu đơn phân tử đã được thu nhỏ đáng kể. Các hệ thống của Oxford Nanopore Technologies như GridION, MinION hoặc Flongle là các hệ thống cầm tay, có thể giải trình tự chuỗi RNA và ADN với lượng đọc dữ liệu lên đến hơn 2 Mb.
Illumina cũng ra mắt series thiết bị Novaseq, đạt đến đỉnh cao mới về sức mạnh giải trình tự cho các nền tảng thương mại. Trên mỗi lần chạy, NovaSeq 6000 (S4 flow cell) tạo đầu ra lên đến 3000 Gb (Illumina, 2017). Các thiết bị đọc dữ liệu dài của Illumina đang được hoàn chỉnh trong tương lai, hứa hẹn tạo nên sự linh hoạt và tăng khả năng mở rộng quy mô, giúp giảm chi phí giải trình tự toàn bộ hệ gene người.
(Nguồn thamm khảo:
https://www.genome.gov/dna-day https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4727787/
https://the-dna-universe.com/2020/11/02/a-journey-through-the-history-of-dna-sequencing/ https://www.pacb.com/blog/the-evolution-of-dna-sequencing-tools/)